미니프로젝트 + 공모전 때문에 제대로 복습하지 못했던 3주..!
너무 블로그가 밀려서 호다닥 업데이트 해야겠다.
담주는 ADsP 시험인데 오또카지 ( ఠ్ఠ ˓̭ ఠ్ఠ )
** 들어가기 전 **
Target이 있는 데이터로 학습하는 지도학습과 다르게, 비지도학습은 Target 변수가 없는 경우에 사용된다.
- 주어진 데이터에서 유사한 특을 가진 데이터 포인트들을 묶어 군집(Cluster)를 형성하는 것
- 학습시 x만 사용하며, 비지도 학습으로 끝나는게 아니라 차원축소, 클러스터링, 이상탐지 등 후속작업이 필요
| [차원축소] | 고차원 데이터를 축소하여 새로운 Feature 형성 => 시각화, 지도학습 연계 |
| [클러스터링] | 고객별 군집 생성 => 고객 집단의 공통 특성 도출을 위한 추가 분석 |
| [이상탐지] | 정상 데이터 범위 지정 => 범위 밖 데이터를 이상치로 판정 |
<차원축소>
1. 차원 축소하는 이유
(1) 희박한(Sparse) 데이터
√ 차원의 수 = 변수의 수

√ 다양한 변수를 고려하여, 모델 성능이 향상
- 고객의 건강상태를 분석할 때, 키/몸무게(2차원) 외 혈압, 체성분 지수, 나이(5차원)으로 하면 더 구체적인 건강상태 분석이 가능
- 그러나 변수가 많아지면, 꼭 필요한 데이터가 아닌데 포함되는 등 불필요하게 복잡해짐
- 변수가 많아질 수록 데이터가 굉장히 희박해짐 (고차원일수록 여러 변수간의 조합에서 비어있는 값이 많음)
(2) 차원의 저주 (Curse of Dimension)
- 따라서, 데이터가 희박해지만 학습이 적절하게 되지 않을 가능성이 높아짐
2. 해결방안
(1) 고차원 문제의 본질 : [희박한 데이터]
- 행을 늘리기 (데이터 늘리기) => 현실적으로 데이터를 늘리는 것은 한계가 있음
- 열을 줄이기 (차원 축소)
3. 차원 축소 (주성분 분석(PCA), t-SNE)
| 특징 | 용도 | 주의사항 |
| 1) 정보유지 - 데이터 중요 정보와 구조를 최대한 유지 - 주성분 분석(PCA) : 데이터 분산 최대화 2) 시각화 - 고차원 데이터를 2차원/3차원으로 축소하여 데이터 분포나 패턴 시각화하여 클러스트 확인할 수 있음 3) 노이즈 제거 - 차원 축소 과정에서 불필요한 변수를 제거하여 데이터 품질 향상 |
1) 데이터 시각화 - 복잡한 데이터 구조 쉽게 이해 - t-SNE는 고차원 데이터 클러스트를 2D 또는 3D로 시각화 2) 노이즈 제거 3) 기계학습 모델 성능 향상 - 과적합 방지 4) 계산 효율성 - 100차원 > 2차원 |
1) 해석의 어려움 - 기존의 변수를 조합하여 만든 새로운 변수로, 해석이 어려움 2) 과적합 - 너무 많은 차원 축소 위험 |
(1) 주성분 분석 PCA
√ 투영
- 정보의 특성을 최대한 유지(정보가 적게 손실)시키며 차원을 축소 => 분산을 최대한 유지
 |
 |
√ PCA 절차

- 데이터 셋 차원만큼의 축을 찾음 (가장 적합한 크기)
- PC1: 데이터의 변동성 중 가장 큰 부분을 설명하는 1차원 축.
- PC2: PC1이 설명하지 못한 나머지 변동성 중 두 번째로 큰 부분을 설명하는 2차원 축.
- PCn: 이후 n번째 주성분 축은 앞의 PC들이 설명하지 못한 나머지 변동성을 설명합니다.
① PCA 사용하기
- 전처리 : 스케일링 필요 ( KNN, SVM...)
- 스케일링 없이 PCA를 수행하면, 스케일이 가장 큰 변수에 영향을 많이 받게 됨
② PCA 문법
# 주성분 분석 선언
pca = PCA(n_components=n)
# 만들고, 적용하기
x_train_pc = pca.fit_transform(x_train)
x_val_pc = pca.transform(x_val)③ 주성분 개수 정하기 (Elbow Method)
# 원본데이터 전체 분산 대비 누적 주성분의 차이 비율
plt.plot(range(1, n+1), pca.explained_variance_ratio_, marker='.')
plt.xlabel('NO. of PC')
plt.grid()
plt.show() |
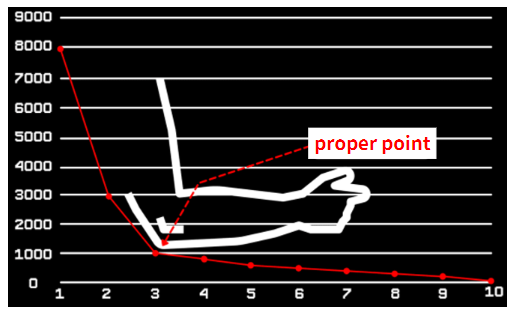 |
* Elbow Method => 팔꿈치 지점 근방에서 적절한 값을 찾는 것
(2) 주성분 PCA 실습
from sklearn.decomposition import PCA
# 주성분 수 2개
n = 2
pca = PCA(n_components = n)
# 만들고, 적용하기(결과는 넘파이 어레이)
x2_pc = pca.fit_transform(x2)
# (옵션) 데이터프레임으로 변환
x2_pc = pd.DataFrame(x2_pc, columns = ['PC1', 'PC2'])
x2_pc.head()
# 두개의 주성분 시각화
sns.scatterplot(x = 'PC1', y = 'PC2', data = x2_pc, hue = y)
plt.grid()
plt.show()

# 주성분 1개 ~ 30개까지 늘려가면서, 그때마다의 성능 확인하기
result = [] # 빈 리스트
for n in range(1,31):
# 데이터 준비
cols = column_names[:n]
x_train_pc_n = x_train_pc.loc[:, cols]
x_val_pc_n = x_val_pc.loc[:, cols]
# 모델링
model = KNeighborsClassifier()
model.fit(x_train_pc_n, y_train)
# 예측
pred = model.predict(x_val_pc_n)
# 평가
result.append(accuracy_score(y_val, pred))
# 시각화
plt.plot(range(1,31), result, marker='.')
plt.axhline(accuracy_score(y_val, pred0), color='r')
plt.grid()
plt.show()
=> 주성분 어케 쓰지?
- 원본데이터와 concat

(3) [추가] t-SNE => 주로 데이터 시각화를 위해 사용됨.(유사도 유지)
- PCA는 선형 축소 방식으로 분산의 크기만 고려하여, 저차원에서 특징을 잘 담아내지 못하는 경우가 발생함
- 원본의 유사도 맵 / 축소된 데이터 유사도 맵의 오차를 줄이는 방향으로 축소된 데이터 조정
from sklearn.manifold import TSNE
# 2차원으로 축소하기
tsne = TSNE(n_components=2)
x_tsne = tsne.fit_transform(x) |
 |
# 좌 : PCA / 우 : tSNE
'머신러닝 > 비지도학습' 카테고리의 다른 글
| [머신러닝(비지도)] #3 - Anomaly Detection (이상 탐지) (2) | 2024.10.22 |
|---|---|
| [머신러닝(비지도)] #2 - Clustering_241021 (0) | 2024.10.21 |

